ROC曲線の書き方を紹介します。RではROC曲線を書くのはとても簡単です。ROC曲線を描くためのライブラリはたくさんありますが、今回はよく使われていて使いやすいpROCライブラリでの書き方を紹介します。EZRなどでも書くことはできますが、自分で書ける方が色々と応用できます。
サンプルデータを作る
ここはサンプルデータを作るだけのところですので、不要な方は次の見出しに読み飛ばしてください。二つの検査、”assay1″と”assay2″でどの程度疾患の人を検出できるのかを検討してみます。
set.seed(3)
condition <- rep(c("healthy", "disease"), each = 50)
assay1 <- c(rnorm(40, mean = 1, sd = 2),
rnorm(10, mean = 3, sd = 1),
rnorm(40, mean = 5, sd = 2),
rnorm(10, mean = 7, sd = 1))
assay2 <- c(rnorm(30, mean = 1, sd = 3),
rnorm(20, mean = 2, sd = 2),
rnorm(30, mean = 3, sd = 3),
rnorm(20, mean = 4, sd = 2))
df = data.frame(condition, assay1, assay2)
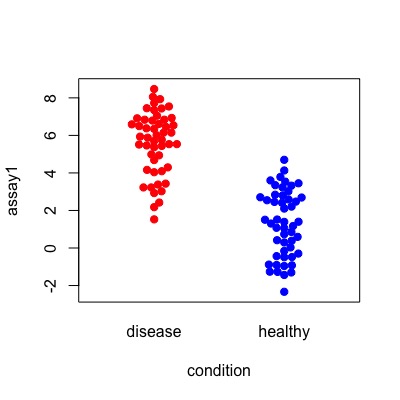
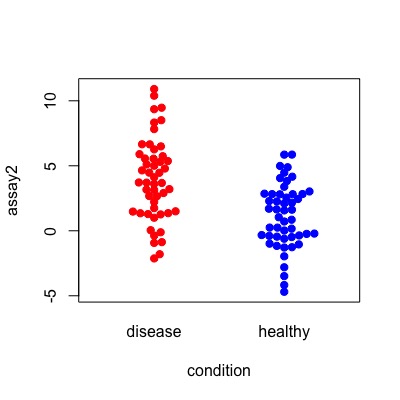
ちなみにこれらの分布はこのようなものになります。
library(beeswarm)
beeswarm(data = df, assay1 ~ condition,
col = c("red","blue"), pch=19)
beeswarm(data = df, assay2 ~ condition,
col = c("red", "blue"), pch=19)
pROCを使ってみる
pROCパッケージのインストール
pROCパッケージをインストールして、起動します。
install.packages("pROC")
library(pROC)
1つのROC曲線を描く
早速いってみましょう。まずデータフレームは次のようになっています。
> head(df)
condition assay1 assay2
1 healthy -0.9238668 2.8520501
2 healthy 0.4149486 -0.2152325
3 healthy 1.5175764 4.1593113
4 healthy -1.3042638 2.8068527
5 healthy 1.3915657 4.0523835
6 healthy 1.0602479 2.8245020
ここからもいくつか方法はありますが、まずroc()を使って、その結果をオブジェクトに格納してみましょう。
roc1 <- roc(condition, assay1, data=df, ci=TRUE,
levels=c("healthy", "disease"))
次のように”~”を使う書き方でも大丈夫です。
roc1 <- roc(df$condition ~ df$assay1, ci=TRUE,
levels=c("healthy", "disease"))
conditionの列は0, 1でもいけますが2値変数です。ciは信頼区間です。
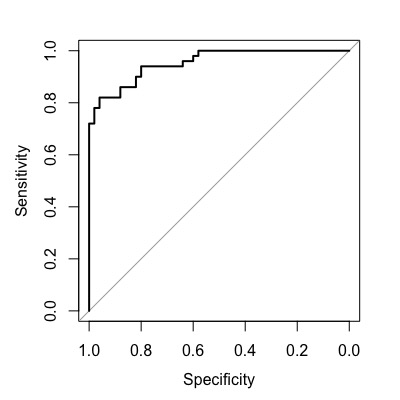
plot()でROC曲線を描くことができます。
plot(roc1)
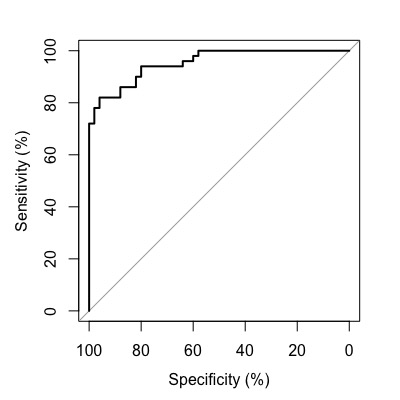
パーセント表示にする
好みにより軸をパーセント表示にすることができます。percent=TRUEを付け加えます。
roc1 <- roc(condition, assay1, data=df, percent=TRUE,
levels=c("healthy", "disease"))
plot(roc1)
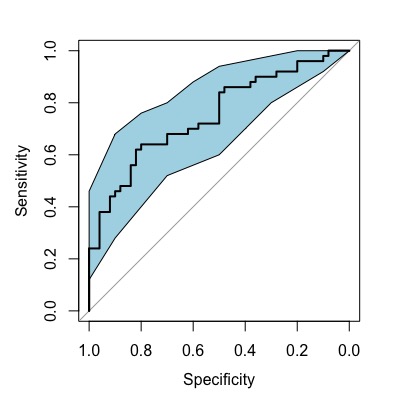
感度の信頼区間を表示する
感度の信頼区間を計算するには ci.se()を使います。colは色を指定します。
roc2 <- roc(condition, assay2, data=df, ci=TRUE,
levels=c("healthy", "disease"))
plot(roc2)
rocCI <- ci.se(roc2)
plot(rocCI, type="shape", col="lightblue")
カットオフに最適な点を表示する
ROC曲線から大事なことは、カットオフに最も適した点を調べることです。最適なカットオフは何か、というところも大きなテーマですが、ここでは2つの方法を紹介します。”Youden Index”と”top left”です。簡単に説明すれば、Youden Indexは”感度+特異度-1” で計算され、その数値が一番高いところを選びます。また、top leftはROC曲線の一番左上に近い点を抽出します。
Youden Indexによる最適なカットオフを表示する
この時にはplotの中に追記をしていきます。print.thres = “best”, print.thres.bes.metod = “youden”とすると最適点が表示されます。また、legacy.axes=TRUEにするとx軸を1-Specificityになります。
plot(roc1, main = "ROC Curve",
identity = TRUE,
print.thres = "best",
print.thres.best.method = "youden",
legacy.axes = TRUE)
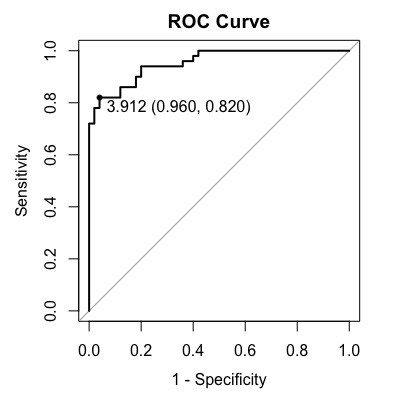
この場合カットオフは3.912で感度が0.820、特異度は0.960です。
Top Leftによる最適なカットオフを表示する
top leftの場合はprint.thres.best.method=”closest.topleft”にします。
plot(roc1, main = "ROC Curve",
identity = TRUE,
print.thres = "best",
print.thres.best.method = "closest.topleft"
legacy.axes = TRUE)
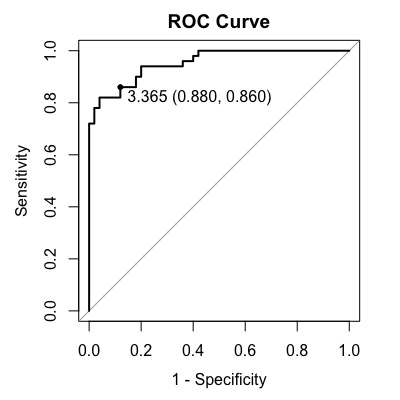
この場合のカットオフは3.365、感度0.860、特異度0.880になっています。括弧内は特異度、感度の順番になっていることに注意をしてください。
必要な数値を取り出す
AUC(area under the curve)の値や感度、特異度などの数値を発表や論文で示す必要があります。その時には次のように表示できます。
> auc(roc1)
Area under the curve: 0.9536
# youden
> coords(roc1, "best", ret=c("threshold", "sens", "spec", "ppv", "npv"))
threshold sensitivity specificity ppv npv
threshold 3.912407 0.82 0.96 0.9534884 0.8421053
# topleft
> coords(roc1, "best", best.method="closest.topleft", ret=c("threshold", "sens", "spec", "ppv", "npv"))
threshold sensitivity specificity ppv npv
threshold 3.364778 0.86 0.88 0.877551 0.8627451
2つのROC曲線を重ねて比較する
今回2つの検査を想定してサンプルデータを作りました。どちらの検査が優れているかみていきます。
2つのROC曲線を重ねる
2つのROC曲線を重ねる時には後から重ねる方をlines()にします。または単純にplot(…, add=TRUE)でも同じ結果になります。colで色を指定しています。
# 2つの検査について、roc()を使ってその結果をオブジェクトに格納する
roc1 <- roc(condition, assay1, data=df, ci=TRUE,
levels=c("healthy", "disease"))
roc2 <- roc(condition, assay2, data=df, ci=TRUE,
levels=c("healthy", "disease"))
# linesを使う
obj1 <- plot(roc1,
col="red")
obj2 <- lines(roc2,
col="blue")
# add = TRUEにする
obj1 <- plot(roc1,
col="red")
obj2 <- plot(roc2,
col="blue",
add=TRUE)
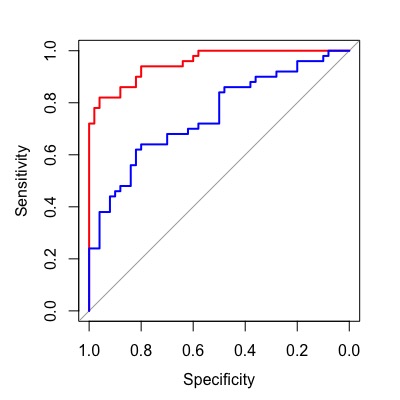
2つの検査について比較する
2つの検査について比較する時にはroc.test()を使います。デフォルトではAUC(area under the curve)をDeLong法で比較しますが、method = “bootstrap”と指定すればブートストラップ法などにも変更できます。textで中央にp値を表示しています。またタイトルはmain=で指定できます。
roc1 <- roc(condition, assay1, data=df, ci=TRUE,
levels=c("healthy", "disease"))
roc2 <- roc(condition, assay2, data=df, ci=TRUE,
levels=c("healthy", "disease"))
obj1 <- plot(roc1,
main="Comparison",
col="red")
obj2 <- lines(roc2,
col="blue")
obj <- roc.test(obj1, obj2)
text(.5, .5, labels=paste("p-value =", format.pval(obj$p.value, 3)),
adj=c(0, .5))
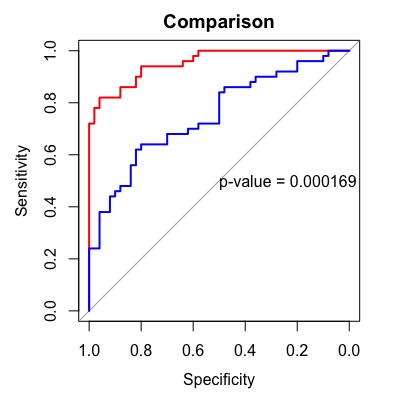
比較の結果を見るだけだったらroc.test()で結果が出てきます。
> roc.test(roc1, roc2)
DeLong's test for two correlated ROC curves
data: roc1 and roc2
Z = 3.7621, p-value = 0.0001685
alternative hypothesis: true difference in AUC is not equal to 0
95 percent confidence interval:
0.0969536 0.3078464
sample estimates:
AUC of roc1 AUC of roc2
0.9536 0.7512
最後にlegendを入れてみます。
roc1 <- roc(condition, assay1, data=df, ci=TRUE,
levels=c("healthy", "disease"))
roc2 <- roc(condition, assay2, data=df, ci=TRUE,
levels=c("healthy", "disease"))
obj1 <- plot(roc1,
main="Comparison",
col="red")
obj2 <- lines(roc2,
col="blue")
obj <- roc.test(obj1, obj2)
# p values in the graph
text(.5, .5, labels=paste("p-value =", format.pval(obj$p.value, 3)),
adj=c(0, .5))
#legend
legend("bottomright", legend=c("Assay1", "Assay2"),
col=c("red", "blue"), lty=1, lwd=2)
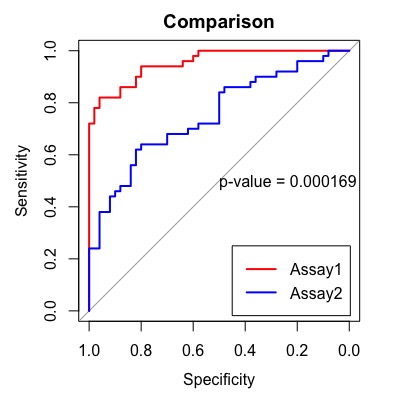
綺麗なROC曲線を書くことができました。
いかがでしたでしょうか。お役に立ちましたら幸いです。